© М.Р. Гайнуллин, А.С. Шорина, 2003 г.
УДК 576.8
Поступила 15.01.2003 г.
М.Р. Гайнуллин, А.С. Шорина
Государственная медицинская академия; ЦНИЛ, Нижний Новгород
Биоинформатический анализ особенностей пространственного строения белков-мишеней убиквитилирования
Биоинформатика — новая междисциплинарная область исследований, развивающаяся на стыке биологии и современных компьютерных технологий. Толчком к ее возникновению послужило, в первую очередь, быстрое накопление экспериментальных данных о структуре биомакромолекул — белков и нуклеиновых кислот. С момента опубликования первых работ о расшифровке аминокислотных и нуклеотидных последовательностей объем такого рода информации растет в экспоненциальной зависимости. В частности, количество материалов о структуре ДНК, депонированных в базе данных европейской молекулярно-биологической лаборатории EMBL Nucleotide Sequence Database, удваивается каждые 12 мес [1, 2]. Другой важнейшей предпосылкой развития биоинформатики стала возможность быстрого и общедоступного обмена информацией с помощью сети Интернет. Потенциал WWW (World Wide Web) позволяет претворить в жизнь новую парадигму современных компьютерных технологий — интеграцию пространственно удаленных систем хранения и обработки информации [3].
Развитие биоинформатических исследований осуществляется, в первую очередь, за счет свободного доступа к специализированным молекулярно-биологическим базам данных. Большинство их содержит исключительно структурные данные, однако в последнее время предпринимаются попытки интегрировать в соответствующих записях также необходимую дополнительную информацию. К примеру, в аннотированной базе данных SWISS-PROT собраны не только сведения о первичных структурах (более чем 120 тыс. белков), но также данные об их функции, особенностях доменной организации, возможных посттрансляционных модификациях и соответствующие литературные ссылки [4, 5]. Особый интерес представляет собой база PDB (Protein Data Bank), в которой депонирована информация о пространственной структуре белков, полученная методами рентгеноструктурного анализа и ядерно-магнитного резонанса [6, 7].
Следует подчеркнуть, что данные, депонированные в соответствующих базах, получены экспериментальным путем, и, следовательно, являются фактическим материалом. Электронная форма представления этих данных позволяет любому исследователю проводить необходимый ему анализ в соответствии с поставленными целями и задачами. Методический арсенал биоинформатики исключительно широк и практически ежедневно пополняется новыми концепциями и алгоритмами. Это позволяет решать большое число конкретных научных задач, используя имеющийся в наличии пакет специализированного программного обеспечения, либо опираясь на возможность интерактивного обращения к соответствующим ресурсам глобальной сети Интернет.
Находясь в процессе непрерывного динамического развития, биоинформатика — как самостоятельная область биохимии и молекулярной биологии — открывает перед исследователями практически неограниченные перспективы для решения фундаментальных и прикладных проблем современной биохимии [8—10]. В нашей работе мы использовали биоинформатические инструменты для исследования системы убиквитина.
Изменение физико-химических свойств и биологической активности белков путем их посттрансляционной модификации является широко распространенной стратегией регуляции метаболических процессов в живой клетке. Один из таких механизмов реализуется за счет связывания (конъюгации) белка-мишени и убиквитина — термостабильного высококонсервативного белка с молекулярной массой 8,5 кДа. Процесс конъюгации заключается в образовании ковалентной изопептидной связи между C-концевым глицином (Gly76) молекулы убиквитина и e-аминогруппой остатка внутреннего лизина полипептидной цепи белка-мишени [11].
Убиквитилирование белков является энзиматическим АТФ-зависимым процессом. Многоступенчатую реакцию катализируют три различных фермента, обозначаемых обычно как компоненты Е1, Е2 и Е3 мультиферментного убиквитин-протеин лигазного комплекса. На первом этапе компонент Е1 (убиквитин-активирующий фермент) с использованием энергии АТФ связывает молекулу убиквитина. После этого происходит перенос убиквитина на компонент Е2, называемый «убиквитинконъюгирующий фермент». Компонент Е3, или собственно убиквитин-протеин лигаза, выполняет функции связывания белка-мишени и переноса молекулы убиквитина на соответствующий целевой внутренний остаток лизина, и, таким образом, завершает весь каталитический акт убиквитилирования. Так как в процессе катализа неизбежно физическое взаимодействие как между компонентами Е2 и Е3, так и между компонентом Е3 и белком-мишенью, компонент Е3 иногда именуется «docking protein»[12]. Схематично последний этап убиквитилирования — ковалентное присоединение молекулы убиквитина к белку-мишени, происходящее при участии компонентов Е2 и Е3 убиквитин-протеин лигазного комплекса, – представлен на рис. 1, а.
Рис. 1. Схема взаимодействия компонентов Е2 и Е3 убиквитин-протеин
лигазного комплекса с убиквитином и белком-мишенью убиквитилирования
при образовании: а — убиквитин-белкового конъюгата 1-го порядка
(моноубиквитилированный белок-мишень); б — убиквитин-белкового
конъюгата 2-го порядка (диубиквитилированный белок-мишень). S —
субстрат убиквитилирования (белок-мишень); Ubi 1 и Ubi 2 – молекулы
убиквитина, последовательно вступающие в реакцию; LysX, Lys48,
Gly76 – аминокислотные остатки белка-мишени и убиквитина, участвующие
в образовании изопептидной связи; волнистой чертой обозначена
тиоэфирная связь между С-концевым глицином (Gly76) молекулы убиквитина
и остатком цистеина активного центра убиквитин-протеин лигазы
|
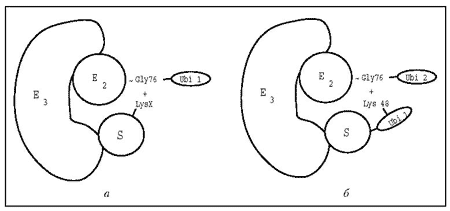 |
Установлена способность убиквитина к полимеризации с образованием «разветвленной» мультиубиквитиновой цепи. Механизм формирования мультимера аналогичен конъюгации с белком-мишенью с тем лишь отличием, что в качестве акцепторной аминокислоты при образовании изопептидной связи выступает остаток лизина 48 (Lys48) другой молекулы убиквитина [13, 14]. Схема удлинения мультиубиквитиновой цепи отображена на рис. 1, б.
Метаболическая роль убиквитилирования внутриклеточных белков является в настоящее время предметом интенсивных исследований. Показано, что субстратами убиквитилирования являются многие белки, значимые в метаболическом отношении [11, 12, 15, 16]. Учитывая критическую роль данной посттрансляционной модификации в регуляции многих важнейших клеточных функций, представляется неоспоримым существование механизма тонкого ферментативного контроля над специфичностью отбора белков-мишеней. Однако, несмотря на значительное количество идентифицированных белков-субстратов убиквитилирования (на сегодняшний день более ста), вопрос о специфичности убиквитин-протеин лигазы остается нерешенным [17]. Значительную сложность данной проблеме придает исключительная гетерогенность пула субстратов убиквитилирования, включающего, в частности, такие разнородные по своим физико-химическим свойствам, структуре и внутриклеточной локализации белки, как гистоны, рецепторы гормонов и факторов роста, ферменты и т.д.
Так как нам представлялась целесообразной разработка методов биоинформатического анализа структурных особенностей белков-субстратов убиквитилирования, то целью данной работы стал поиск подходов к решению вопроса о субстратной специфичности убиквитин-протеин лигазы путем выявления сходства в пространственной структуре молекулы убиквитина и идентифицированных субстратов убиквитилирования. Поставленная задача решалась биоинформатическими методами с использованием информации о третичной и четвертичной структуре белков, депонированной в базе данных RCSB Protein Data Bank, пакета специализированных прикладных программ для визуализации и анализа пространственной организации биомакромолекул и оригинальной компьютерной программы, служащей для расчета расстояний между определенными аминокислотами в трехмерных структурах исследуемых белков.
Методы исследования.
Выбор объектов для биоинформатического анализа. Отбор белков-мишеней проводился с учетом следующих критериев: 1) наличие прямых экспериментальных данных об убиквитилировании данного белка (включение радиоактивно меченного убиквитина в состав конъюгата в опытах in vitro, результаты иммунохимического анализа или определение аминокислотной последовательности in vivo); 2) доступность трехмерной структуры целого белка (наличие кристаллографических данных, депонированных в базе данных PDB). Дополнительным критерием отбора служило наличие информации о специфическом убиквитилировании определенного остатка лизина.
Исходные данные. Для анализа использовалась информация о третичной структуре белков, депонированная в базе данных PDB под кодами доступа 1UBQ [18], 1EQZ [19] и 1F6S [20] для убиквитина, гистона H2A и a-лактальбумина соответственно.
Программное обеспечение. Визуализация трехмерной структуры белка, выявление аминокислот, формирующих поверхность молекулы, и расчет расстояния между ними выполнялись с помощью программ RasWin Molecular Graphics 2.7.1 [21] и Accelrys ViewerPro 4.2. Оценка расположения отдельных аминокислотных остатков на поверхности белковой глобулы проводилась по степени их экспонированности в окружающую среду. Для этого использовалась программа MOLMOL, позволяющая проводить расчет доступности для растворителя аминокислотных остатков и отдельных функциональных групп [22]. Во всех случаях оценка экспонированности проводилась при стандартном радиусе молекулы растворителя, равном 1,4Е. Для исследования пространственного взаиморасположения определенных аминокислот применялась оригинальная программа Distance Searcher, написанная на языке C++ (разработчик — С.В. Припоров). Внешний вид интерфейса программы представлен на рис. 2.
| Рис. 2. Внешний вид интерфейса программы Distance Searcher
|
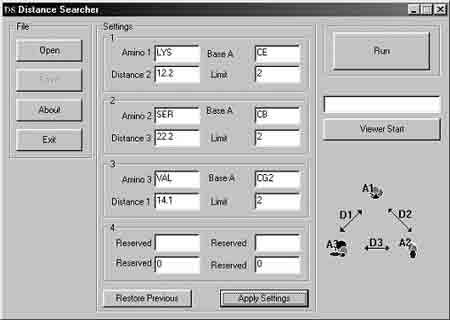 |
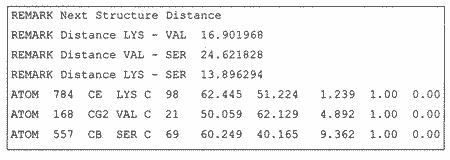
Данная программа позволяет выявлять аминокислоты, находящиеся на заданном расстоянии друг от друга. Алгоритм поиска основан на анализе координат атомов, содержащихся в файле PDB. Пользователь определяет искомые аминокислоты (максимально четыре), задает дистанцию в Е между определенными атомами, входящими в их состав, и величину допустимого отклонения от этого расстояния. Результаты анализа сохраняются в текстовом файле формата PDB и содержат информацию о наборе аминокислот, удовлетворяющих заданным требованиям, выявленные дистанции между парами аминокислот и фрагменты из исходного файла данных, характеризующие каждую найденную аминокислоту (рис. 3).
Рис. 3. Результат поиска потенциальных точек убиквитилирования
в трехмерной структуре a-лактальбумина, выполненного с помощью
программы Distance Searcher (фрагмент файла отчета)
|
 |
Результаты. Теоретической основой работы служило предположение, что убиквитин-протеин лигаза способна специфически распознавать и взаимодействовать с аминокислотами, формирующими поверхность молекулы белка-мишени вокруг точки убиквитилирования — остатка лизина, участвующего в образовании изопептидной связи с молекулой убиквитина. Это предположение основано на том, что в процессе ферментативного удлинения мультиубиквитиновой цепи изопептидная связь формируется при участии лишь одного из семи остатков лизина, имеющихся в составе молекулы убиквитина, а именно Lys48. Исходя из того, что обе стадии формирования убиквитин-белкового конъюгата — убиквитилирование белка-мишени и наращивание мультиубиквитиновой цепи — катализируются теми же компонентами убиквитин-протеин лигазного комплекса, можно допустить, что поверхности обеих белковых молекул содержат идентичные аминокислоты, по которым и происходит специфическое «узнавание» и связывание субстрата ферментом (см. рис. 1).
Основываясь на указанных критериях отбора, помимо собственно убиквитина для биоинформатического анализа нами были взяты молекулы гистона Н2А и a-лактальбумина. Гистон Н2А – исторически первый белок, для которого показана возможность убиквитилирования in vivo и in vitro [23, 24]. Механизм его конъюгации с убиквитином детально охарактеризован, включая идентификацию остатка лизина 119 (Lys119), специфически участвующего в образовании изопептидной связи. Молекула a-лактальбумина, в свою очередь, служит популярным модельным субстратом в экспериментальных исследованиях процесса убиквитилирования [25]. Тот факт, что a-лактальбумин не является нативным эндогенным белком-мишенью, позволяет упростить интерпретацию результатов биоинформатического анализа за счет исключения возможной регуляторной роли факторов более высокого функционального уровня на каталитический процесс.
Проведен анализ структурных предпосылок специфического характера участия остатка Lys48 в образовании изопептидной связи при формировании мультиубиквитиновой цепи. Для исключения фактора возможных стерических ограничений рассчитана степень экспонированности для всех семи остатков лизина, присутствующих в молекуле убиквитина: Lys6, Lys11, Lys27, Lys29, Lys33, Lys48, Lys63. Установлено, что доступность для растворителя e-аминогрупп всех указанных аминокислот, за исключением остатка Lys27, колеблется в пределах от 50 до 90%. Эти результаты указывают на то, что специфичность убиквитилирования Lys48 определяется ферментативным механизмом.
На следующем этапе работы нами был проведен анализ поверхности молекулы убиквитина вокруг остатка Lys48 и поверхности молекулы гистона H2A вокруг остатка Lys119. Визуализация трехмерных структур этих белков с помощью программ RasWin и ViewerPro дала возможность проанализировать и сравнить аминокислотный состав, формирующий поверхность молекул этих белков в выбранной области. Сравнение аминокислотного окружения точек убиквитилирования выявило в обеих белках элементы подобия. Оба белка содержат остатки валина и серина, сходным образом позиционированные вокруг соответствующего остатка лизина. Это валин 70 (Val70), серин 57 (Ser57) и валин 114 (Val114), серин 122 (Ser122) для молекул убиквитина и гистона H2A соответственно (рис. 4).
Рис. 4. Трехмерные структуры убиквитина (а), гистона Н2А (б)
и a-лактальбумина (в) и локализация идентично позиционированных
остатков лизина, валина и серина на поверхности белковых молекул
|
С помощью программы для анализа трехмерных структур ViewerPro, были рассчитаны дистанции между идентичными парами аминокислот в исследуемых белках. Расчет дистанций в обоих случаях дал близкие значения: Lys—Val — 14±1Е, Lys—Ser — 12±1Е и Ser—Val — 22±2Е (см. таблицу).
Молекулярные дистанции (в Е) между идентичными парами
аминокислот в исследованных белках
|
 |
С помощью программы Distance Searcher в молекуле a-лактальбумина проведен поиск аминокислот, отвечающих выявленным критериям. Установлено, что заданные стерические закономерности выполняются для группы лизин-валин-серин, включающей Lys98, Val21 и Ser69 (см. таблицу и рис. 4). Учитывая тот факт, что для a-лактальбумина точка убиквитилирования не идентифицирована, проведенный анализ можно рассматривать как инструмент предсказания акцепторного лизина. Полученные результаты позволяют предположить, что в убиквитилировании a-лактальбумина принимает участие остаток Lys98.
Обсуждение. Метаболическая значимость системы убиквитина определяется ее вовлечением в регуляцию ряда важнейших клеточных процессов: экспрессии генов, апоптоза, передачи внутриклеточного сигнала, пострепликативной репарации ДНК и многих других. Особый интерес для медицины представляют данные о роли убиквитина в патогенезе нейродегенеративных заболеваний, ишемии мозга и в механизмах канцерогенеза [26—29], вследствие чего система убиквитина в последние годы является предметом интенсивных исследований. Однако несмотря на значительный прогресс в изучении энзимологической стороны проблемы, многие аспекты функционирования данной метаболической системы остаются невыясненными. Одной из актуальных проблем в этой области является установление механизма распознавания ферментом белков-субстратов.
На сегодняшний день предложено несколько возможных механизмов, детерминирующих субстратную специфичность фермента по отношению к различным группам белков-мишеней [15, 30]. Однако ни один из них не является универсальным, что может быть связано со значительной гетерогенностью пула белков-субстратов и, как следствие, многообразием метаболических эффектов убиквитилирования. Накопленные экспериментальные данные дают основание предположить, что механизм специфичности убиквитин-протеин лигазы может быть не уникален для разных групп субстратов.
Накопленный объем функциональных и структурных данных делает целесообразным использование инструментов биоинформатического анализа для исследования механизма, определяющего специфичность отбора субстратов убиквитилирования. Исходя из того, что сравнение первичных структур белков-мишеней на сегодняшний день не принесло положительных результатов [17], в наших исследованиях был сделан акцент на изучении пространственной организации субстратов убиквитилирования и их сравнении с трехмерной структурой поверхности убиквитина. Данный подход основывался на экспериментальных данных об особенностях ферментативного механизма образования убиквитин белковых конъюгатов и удлинения мультиубиквитиновой цепи.
Специфическое распознавание ферментом определенного остатка лизина в молекулах убиквитина и гистона Н2А (Lys48 и Lys119 соответственно) дало нам возможность вычленить на поверхности исследуемого белка ограниченную зону, потенциально ответственную за физическое взаимодействие с молекулой фермента. Наличие у обеих молекул в этой зоне остатков серина и валина, сходным образом позиционированных вокруг соответствующего акцепторного лизина, позволило задать стерические условия для анализа трехмерной структуры следующего белка – a-лактальбумина, для которого специфическая точка убиквитилирования не идентифицирована, и провести идентификацию соответствующих аминокислотных остатков на его поверхности.
Таким образом, выбранная стратегия биоинформатического поиска позволила выявить общие структурные особенности в исследованных белках и получить свидетельства в пользу предложенной нами гипотезы о роли идентичных аминокислотных остатков, локализованных на поверхности белковой молекулы, как специфических элементов, детерминирующих «узнавание» убиквитин-протеин лигазой соответствующих белковых субстратов.
Заключение. Проведенный биоинформатический анализ пространственной организации убиквитина и гистона H2A выявил наличие в обеих молекулах идентичных аминокислотных остатков серина и валина, сходным образом позиционированных вокруг остатка лизина, участвующего в образовании изопептидной связи. С использованием оригинальной компьютерной программы «Distance Searcher» установлено, что поверхность молекулы a-лактальбумина содержит тот же набор аминокислот, отвечающий заданным стерическим условиям. Полученные данные свидетельствуют в пользу предложенной нами гипотезы о роли специфических особенностей трехмерной структуры белков-субстратов в их «узнавании» убиквитин-протеин лигазой. Разработанный нами биоинформатический метод структурного анализа может быть использован для предсказания возможной точки убиквитилирования в белках-мишенях и служить основой экспериментального изучения механизма субстратной специфичности убиквитин-протеин лигазы.
Литература
- Stoesser G., Baker W., van den Broek A., Garcia-Pastor M., Kanz C., Kulikova T. et al. The EMBL Nucleotide Sequence Database: major new developments. Nucl Acids Res 2003; 31: 17—22.
- The EMBL Nucleotide Sequence Database. http://www.ebi.ac.uk/embl/
- Bioanalytik. Lottspeich F., Zobras H. (editors). 1998. Heidelberg; Berlin: Spektrum, Akad. Verl., p. 781—784.
- The SWISS-PROT protein knowledgebase and its supplement TrEMBL. http://www.expasy.org/sprot/
- Appel R.D., Bairoch A., Hochstrasser D.F. A new generation of information retrieval tools for biologists: the example of the ExPASy WWW server. Trends Biochem Sci 1994; 19: 258—260.
- Research Collaboratory for Structural Bioinformatics (RCSB). Protein Data Bank (PDB). http://www.rcsb.org/pdb/
- Berman H.M., Westbrook J., Feng Z., Gilliland G., Bhat T.N., Weissig H. et al. The Protein Data Bank. Nucleic Acids Res 2000; 28: 235—242.
- Дубанов А.В., Иванов А.С., Арчаков А.И. Компьютерный поиск новых белков-мишеней для действия противомикробных средств на основе сравнительного анализа геномов. Вопросы мед химии 2001; 47: 353—367.
- Ivanov A.S., Dubanov A.V., Skvortsov V.S., Archakov A.I. Computer drug design based on analysis of a target macromolecule structure. I. Search and description of a ligand binding site in a target molecule. Vopr Med Khim 2002; 48: 304—315.
- Арчаков А.И. Биоинформатика, геномика и протеомика — науки о жизни XXI столетия. Вопросы мед химии 2000; 46: 4—7.
- Hochstrasser M. Ubiquitin, proteasomes, and the regulation of intracellular protein degradation. Curr Opin Cell Biol 1995; 7: 215—223.
- Pickart C.M. Mechanisms underlying ubiquitination. Annu Rev Biochem 2001; 70: 503—533.
- Van Nocker S., Vierstra R.D. Multiubiquitin chains linked through lysine 48 are abundant in vivo and are competent intermediates in the ubiquitin proteolytic pathway. J Biol Chem 1993; 268: 24766—24773.
- Chau V., Tobias J.W., Bachmair A., Marriott D., Ecker D.J., Gonda D.K. et al. A multiubiquitin chain is confined to specific lysine in a targeted short-lived protein. Science 1989; 243: 1576—1583.
- Weissman A.M. Themes and variations on ubiquitylation. Nat Rev Mol Cell Biol 2001; 2: 169—178.
- Jennissen H.P. Ubiquitin and the enigma of intracellular protein degradation. Eur J Biochem 1995; 231: 1—30.
- Hochstrasser M. New structural clues to substrate specificity in the «ubiquitin system». Mol Cell 2002; 9: 453—454.
- Vijay-Kumar S., Bugg C.E., Cook W.J. Structure of ubiquitin refined at 1.8 A resolution. J Mol Biol 1987; 194: 531—544.
- Harp J.M., Hanson B.L., Timm D.E., Bunick G.J. Asymmetries in the nucleosome core particle at 2.5 A resolution. Acta Crystallogr D Biol Crystallogr 2000; 56(12): 1513—1534.
- Chrysina E.D., Brew K., Acharya K.R. Crystal structures of apo- and holo-bovine alpha-lactalbumin at 2.2-A resolution reveal an effect of calcium on inter-lobe interactions. J Biol Chem 2000; 275: 37021—37029.
- Sayle R.A., Milner-White E.J. RASMOL: biomolecular graphics for all. Trends Biochem Sci 1995; 20: 374.
- Koradi R., Billeter M., Wuthrich K. MOLMOL: a program for display and analysis of macromolecular structures. J Mol Graph 1996; 14: 51—32.
- Goldknopf I.L., French M.F., Musso R., Busch H. Presence of protein A24 in rat liver nucleosomes. Proc Natl Acad Sci USA 1977; 74: 5492—5495.
- Goldknopf I.L., Busch H. Isopeptide linkage between nonhistone and histone 2A polypeptides of chromosomal conjugate-protein A24. Proc Natl Acad Sci USA 1977; 74: 864—868.
- Ferber S., Ciechanover A. Transfer RNA is required for conjugation of ubiquitin to selective substrates of the ubiquitin- and ATP-dependent proteolytic system. J Biol Chem 1986; 261: 3128—3134.
- Scheffner M., Werness B.A., Huibregtse J.M., Levine A.J., Howley P.M. The E6 oncoprotein encoded by human papillomavirus types 16 and 18 promotes the degradation of p53. Cell 1990; 63: 1129—1136.
- Alves-Rodrigues A., Gregori L., Figueiredo-Pereira M.E. Ubiquitin, cellular inclusions and their role in neurodegeneration. Trends Neurosci 1998; 21: 516—520.
- Dewar D., Graham D.I., Teasdale G.M., McCulloch J. Cerebral ischemia induces alterations in tau and ubiquitin proteins. Dementia 1994; 5: 168—173.
- Mayer R.J., Arnold J., Laszlo L., Landon M., Lowe J. Ubiquitin in health and disease. Biochim Biophys Acta 1991; 1089: 141—157.
- Laney J.D., Hochstrasser M. Substrate targeting in the ubiquitin system. Cell 1999; 97: 427—430.
Вверх | Назад
Главная |
Врачам |
Пациентам |
Студентам |
Мед.учреждения |
Мед.анекдоты |
Полезные ссылки
Нижегородский медицинский сайт
по вопросам размещения рекламы пишите здесь
| |

